| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | ||
| 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 13 | 14 | 15 | 16 | 17 | 18 | 19 |
| 20 | 21 | 22 | 23 | 24 | 25 | 26 |
| 27 | 28 | 29 | 30 |
Tags
- Batch effect
- 싱글셀 분석
- single cell
- DataFrame
- scRNAseq analysis
- python matplotlib
- ngs
- single cell analysis
- ChIPseq
- julia
- MACS2
- scRNAseq
- EdgeR
- HTML
- matplotlib
- single cell rnaseq
- 비타민 C
- drug muggers
- cellranger
- github
- CUT&RUN
- CUTandRUN
- CSS
- Bioinformatics
- javascript
- Git
- PYTHON
- pandas
- js
- drug development
Archives
- Today
- Total
바이오 대표
[NGS scRNAseq] cellranger count 의 output 파일, summary.html 해석 본문
Bioinformatics/Tools
[NGS scRNAseq] cellranger count 의 output 파일, summary.html 해석
바이오 대표 2022. 12. 19. 09:44
Cell Ranger
OUTPUT FILES
Cell ranger 의 output 은 outs/ 폴더에 저장이 된다. 해당 폴더에는 sequencing data, the annotated read sequences, gene expression matrices 등이 존재한다.

각 파일에 대한 더 자세한 정보
Summary.html
WEL SUMMARY .html
cell ranger 가 제공해주는 summary 와 analysis 를 html 형식으로 확인할 수 있다. 다음 figure에서 볼 수 있듯이, 크게 summary 와 gene expression 탭으로 분리가 된다.


해당 html를 통하여, 일차적으로 데이터의 quality를 확인할 수 있다. 많은 카테고리 중 기본적으로 주의깊게 봐야 할 카테고리와 해당 기준점은 다음과 같다.
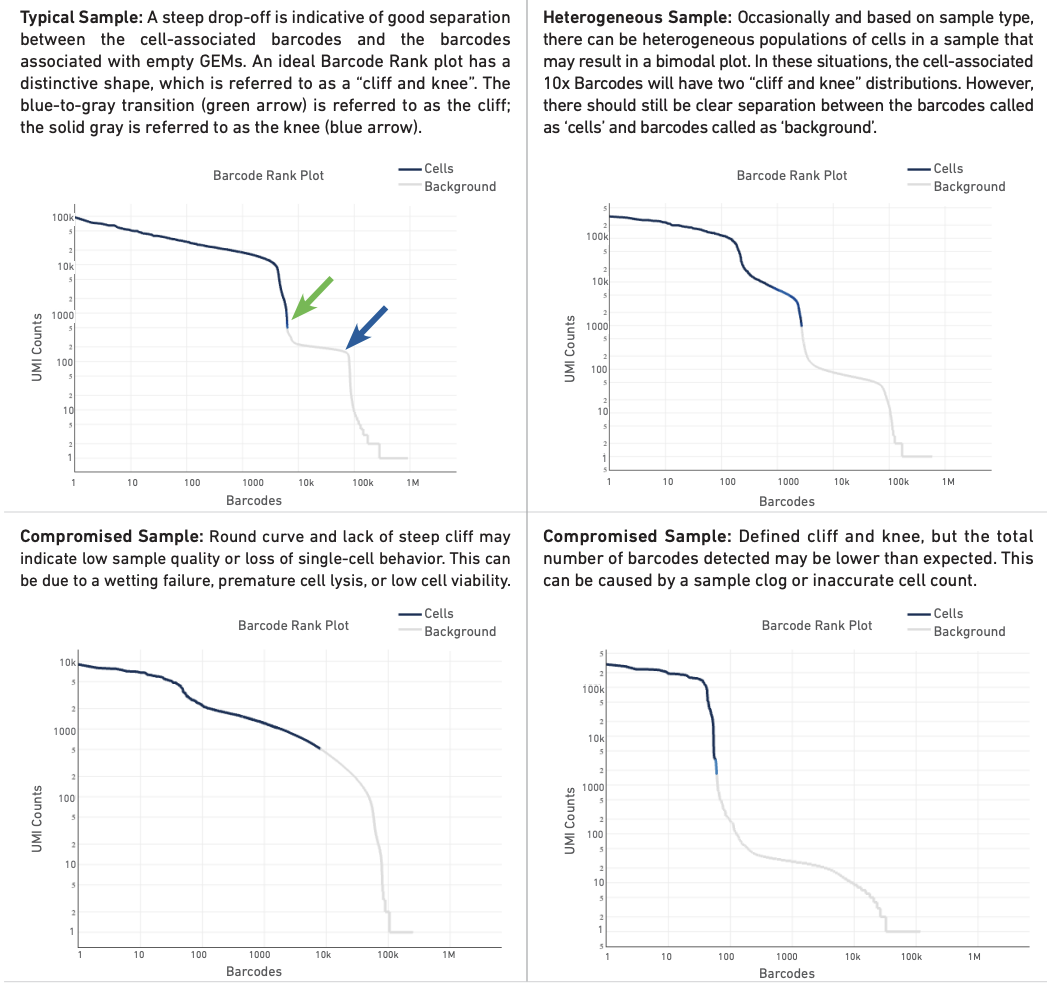
- (Cells) Barcode Rank Plot: 해당 plot 은 barcode counts distribution을 보여준다. steep drop-off 가 good separation (cell-associated barcodes and barcodes associated with empty partitions) 을 의미한다.
- (Cells) Estimated Number of Cells: 500-10,000
- (Cells) Mean Reads per Cell: 20,000 reads/cell, minimum recommended reads per cell around 10,000
- (Sequencing) Valid barcodes: > 75%
- (Sequencing) Q30 bases in RNA read: ideally > 65%6
- (Mapping) Reads mapped confidently to transcriptome: ideally > 30%
- (Mapping) Reads mapped antisense to gene: ideally < 10%
** 보다 자세히 이용하고싶다면, 각 카테고리의 ok 기준점을 확인하고싶다면 https://assets.ctfassets.net/an68im79xiti/163qWiQBTVi2YLbskJphQX/e90bb82151b1cdab6d7e9b6c845e6130/CG000329_TechnicalNote_InterpretingCellRangerWebSummaryFiles_RevA.pdf 를 참고하길 바란다. plot 관련되 설명도 아주 자세히 나와있다.
예시) Bacrdoe Rank Plot 해석하기
'Bioinformatics > Tools' 카테고리의 다른 글
| [ Cut & Tag / Cut & Run ] Cut & Tag 투토리얼 (0) | 2023.03.18 |
|---|---|
| [ NSG QC / trimming ] TrimGalore (0) | 2023.02.14 |
| [ 싱글셀 분석 ] cellranger-atac aggr (0) | 2023.02.03 |
| [NGS scATACseq] scATACseq, Cicero를 이용해서 cis-regulatory gene network 분석하기 (0) | 2022.12.28 |
| [NGS scRNAseq] Chromium 10x Illumina의 기본이해(workflow)와 Cell ranger count (0) | 2022.12.18 |


