| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | ||
| 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 13 | 14 | 15 | 16 | 17 | 18 | 19 |
| 20 | 21 | 22 | 23 | 24 | 25 | 26 |
| 27 | 28 | 29 | 30 |
- drug muggers
- single cell rnaseq
- julia
- CUT&RUN
- PYTHON
- DataFrame
- python matplotlib
- MACS2
- CUTandRUN
- single cell analysis
- 싱글셀 분석
- matplotlib
- Batch effect
- cellranger
- ChIPseq
- ngs
- 비타민 C
- Bioinformatics
- scRNAseq analysis
- Git
- EdgeR
- HTML
- drug development
- CSS
- scRNAseq
- single cell
- github
- pandas
- javascript
- js
- Today
- Total
목록single cell (3)
바이오 대표
 [싱글셀 multiome 논문)“Dictionary learning for integrative, multimodal and scalable single-cell analysis” 2023
[싱글셀 multiome 논문)“Dictionary learning for integrative, multimodal and scalable single-cell analysis” 2023
요약 보통 reference cell type annotation은 scRNAseq의 gene expression 을 기반으로 만들어졌다. 따라서 새로운 데이터의 scRNAseq을 annotate하는 것은 어느정도 쉽지만 다른 modality 갖는 데이터, 즉 gene expression이 아닌 정보를 갖는 데이터 (scATACseq, scCUT&Tag, CyTOF-protein)를 annotate하는데는 어려움이 있다. 따라서 해당 논문에서는 “dictionary learning” 기술을 이용한다. 쉽게 설명하자면 예를 들어, [1] RNA+ATAC정보를 담고있는 10x multiome 을 bridge로 이용하는 것이다. 현존하는 [2] RNA annotated reference 를 [1]scRNA+s..
Abstract 10x Genomics (droplet-based system) 3’ mRNA counting of tens of thousands of single-cell per sample 8 samples at a time 50% cell capture efficiency ~ 250k single cells across 29 samples 로 [1] sensitivity, [2] ability to detect rare population를 파악하고, 68k PBMC cells을 이용하여 [3] ability to characterize large (immune) population 를 실험하였다. 또한 골수 이식환자 데이터의 sequence variation을 이용하여 [4] host/donor..
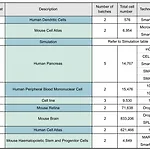 [scRNAseq 논문] 싱글셀 batch integration “A benchmark of batch-effect correction methods for single-cell RNA sequencing data” 2020
[scRNAseq 논문] 싱글셀 batch integration “A benchmark of batch-effect correction methods for single-cell RNA sequencing data” 2020
Abstract 해당 논문에서는 현존하는 14개의 batch correction 방법들을 비교분석한 결과를 다음과 같은 3개의 카테고리에서 보여준다. [1] computational runtime, [2] 큰 데이터셋을 다루는 능력치 [3] 순수 cell type 을 잘 보존하면서 batch correction 을 하는지. 결론적으로는 Harmony, LIGER, Seurat 3 을 추천한다. Introduction 현재 single cell 데이터 batch correction 에 주로 사용되는 알고리즘들을 소개한다. MNNs (Haghverdi et al.) 합치고 싶은 2개의 데이터에서 mutual nearest neighbors (MNNs) 를 계산한다. 그리고 해당 MNNs (list of pa..
