| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | |
| 7 | 8 | 9 | 10 | 11 | 12 | 13 |
| 14 | 15 | 16 | 17 | 18 | 19 | 20 |
| 21 | 22 | 23 | 24 | 25 | 26 | 27 |
| 28 | 29 | 30 | 31 |
- CUTandRUN
- 싱글셀 분석
- MACS2
- drug development
- scRNAseq analysis
- javascript
- matplotlib
- ngs
- Batch effect
- scRNAseq
- 비타민 C
- CSS
- CUT&RUN
- python matplotlib
- cellranger
- single cell rnaseq
- HTML
- github
- Git
- single cell
- EdgeR
- js
- Bioinformatics
- DataFrame
- PYTHON
- pandas
- drug muggers
- ChIPseq
- single cell analysis
- julia
- Today
- Total
바이오 대표
[ Bioinformatics 논문 ] "An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites" 본문
[ Bioinformatics 논문 ] "An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites"
바이오 대표 2022. 9. 27. 06:55
"An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites"
Skene and Henikoff Jan, 2017
Abstract
Cleavage Under Targets and Release Using Nuclease (CUT & RUN)
Cut & Run 역할:
- quantitative high-resolution chromatin mapping
- probing(자세히 조사) of the local chromatin environment
장점: Simple, robust, need ~1/10th the sequencing depth as ChIP (low background data)
따라서 in situ mapping of protein-DNA interaction 에 Chip-seq 대신 CUT&RUN 이 사용 가능하다.
eLife digest
Transcription factors (TFs) 라고 불리는 단백질은 DNA에 붙어서 주의에 있는 유전체들을 On (발현시키거나) / Off (불발) 한다. TFs는 histone과 같은 프로틴과도 interact 한다. 이러한 TFs, histones 혹은 chromatin과 연관된 다른 프로틴들은 모두 nearby gene의 active 조절을 한다.
어떠한 TFs나 chromatin-associated protein 이 DNA의 잘못된 부분에 붙게되면, 인간의 몸에서 질병을 일으킬 수 있다. 암 또한 이중 하나이다. 이러한 이유 때문에 많은 Researcher들이 다양한 상황에서의 specific DNA-binding proteins의 위치를 찾는데 많은 흥미를 갖는다. chromatin immunoprecipitation (ChIP)이라 불리는 기술은 이를 할 수 있게 해주었고 널리 사용되었지만 꽤 문제점이 있었기에 대안방안을 찾았다. 그것이 바로 CUT&RUN (cleavage under targets & release using nuclease) 방법이다 .
CUT&RUN compare to ChIP
장점 1. ChIP과 다르게 DNA가 있는 cells을 처음에 부수지 않아도 된다. 이는 즉 protein-DNA Interaction의 natural state가 보존된다 말할 수 있다.
장점 2: antibody binds to protein interest in intact (온전한) cells, cuts out the DNA --> releasing the DNA fragment from cells
장점 3: 적은 양의 cell로 실험을 시작할수 있다.
Introduction
Chip-seq -> TFs profiling (transcription factors 의 위치 파악)
X-ChIP (formaldehyde crosslinking ChIP), and with ChIP-seq, base-pair resolution mapping of TFs became feasible (Rhee and Pugh, 2011; Skene and Henikoff, 2015; He et al., 2015).
Chip-seq (1980s)
: Chromatin Immunoprecipitation
역할: protein interacted DNA sequencing = cofactor, histon localizing
단점: 대량의 sample이 필요하다. chromatin을 sonication으로 랜덤하게 절단해서 target DNA 부분도 절단되어버리는 경우가 있다.
ATAC-seq (2013) Epigenetics 정보 얻기 - DNA methylation, histone 변형 (acetylation), noncoding-RNA
: Assay for Transposase Accessible Chromatin with high-throughput sequencing
https://www.abpbio.com/product/tn5-transposase/
Tn5 Transposase | ABP Biosciences
Introduction ABPNGS Tn5 Transposase is a hyperactive form of Tn5 transposase. This enzyme can be used to randomly insert Tn5 Transposon into any target DNA, in vitro. In addition to Tn5 Transposase, efficient transposition requires that each Tn5 Transposo
www.abpbio.com
=> chromatin accessibility: tn5 transposage(인공합성) 을 이용하여 Euchraomatic (open) chromatin 에 adaptor을 붙이고 sequencing.
장점: fragmentation, library(adopter 붙이는 것) 를 한번에 실행
Cut & Run (2017)
Cut & Tag (2019)
장점:
[1] 5,000~ 50,000 cells 적은 sample로 실험 가능
[2] transposage가 target protein에 붙어서 그 근방만 절단 -> Low background data 가능하다.

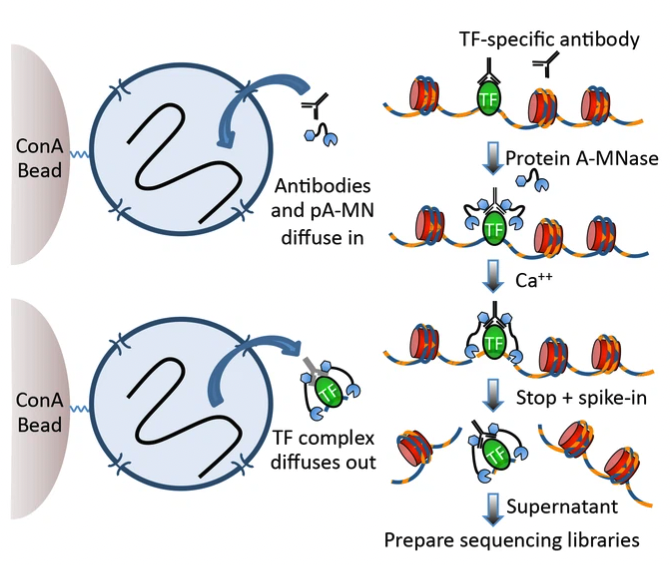
Results
- Cut&RUN produces limit digestion of chromatin complexes
Total DNA extraction ~ purification of small fragments
- Cut&RUN robustly maps yeast TF binding sites in situ at high resolution
In situ research involves removing cells, hormones, or proteins from a living organism and then preserving them to be studied in a laboratory environment.
- CUT&RUN precisely maps chromatin-associated complexes
- CUT&RUN resolves rare insoluble DNA-binding protein complexes
- CUT&RUN probes nearby chromatin
- CUT&RUN maps human transcription factor binding sites at high resolution
- CUT&RUN maps histone modifications in compated chromatin
- CUT&RUN directionally maps long-range genomic contacts
- CUT&RUN allows quantitative measurements with low cell numbers




