| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | ||
| 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 13 | 14 | 15 | 16 | 17 | 18 | 19 |
| 20 | 21 | 22 | 23 | 24 | 25 | 26 |
| 27 | 28 | 29 | 30 |
- Batch effect
- single cell
- ChIPseq
- matplotlib
- HTML
- single cell analysis
- drug development
- pandas
- python matplotlib
- DataFrame
- CUTandRUN
- CSS
- Git
- scRNAseq
- PYTHON
- js
- ngs
- CUT&RUN
- 비타민 C
- 싱글셀 분석
- EdgeR
- single cell rnaseq
- drug muggers
- MACS2
- cellranger
- github
- scRNAseq analysis
- Bioinformatics
- javascript
- julia
- Today
- Total
바이오 대표
[ Bioinformatics 논문 ] 유전자 코드 해석에 NLP 사용 "pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome" 본문
[ Bioinformatics 논문 ] 유전자 코드 해석에 NLP 사용 "pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome"
바이오 대표 2022. 8. 9. 16:41
"pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome"
Feb 2021
Abstract
(해당 논문은 가짜연구소에서 알게된 지인의 관심사로 인해 읽어보았다. sequencing 이후에 서열 해석을 위해 NLP를 사용하는 것이다.)
Motivation: non-coding DNA 를 해독하는 것은 유전체 연구에서 주 문제중 하나이다. Gene regulatory code (유전자 조절 코드)는 gene 의 여러 방면으로 해석이 가능하고 (polysemy), 멀리 떨어진 유전자와도 연관성이 있어 (distant semantic relation ship) 같이 아주 복잡하다. 이런한 복잡도는 이전의 informatics 방법으로는 데이터 부족으로 인해 종종 실패했다.
Results: 위와 같은 문제에 맞서, 우리는 새로운 pre-trained bidirectional encoder representation , DNABERT 을 개발하였다. 이는 up/downstream nucleotide 를 베이스로 타겟 유전자 DNA 서열의 global 하고 transferrable 한 부분을 이해할수 있도록 한다. 우리는 DNABERT 를 genome-wide regulatroy elements prediction 에서 가장 널리 사용되는 프로그램과 사용법이 쉬운지의 접근성과, 정확도와 효과를 비교했다. 우리는 하나의 pre-trained transformers 모델이 약간의 tuning 과 라벨링 후에, promoters, splice sites, 그리고 transcription factor binidng sites 예측 부분에서 현존하고 있는 최고 성능에 도달하였다. 게다가, DNABERT는 각 nucleotide-level의 중요도와 의미관계를 직접적으로 시각화할 수 있고, 이는 더 나은 해석력을 제공하고 conserved sequence motifs 와 functional genetic variant 후보군들을 정확하게 찾을 수 있다. 마지막으로, 인간 게놈으로 pre-trained한 DNABERT모델을 다른 개체에 적용 했을 때도 좋은 성능을 보였다. 우리는 이런 pre-trained DNABERT 모델이 많은 다른 서열 분석 작업에서도 좋을 역할을 해낼 것이라고 예상한다.
1. Introduction
DNA code 해석 -> DNA 가 proteins 으로 어떻게 바뀌는지 설명할 수 있다. 어떠한 cell과 organisms에서 언제, 어떻게 발현되는지 알 수 있다.
같은 cis-regulatory elements (CREs)*는 다른 생물학적 조건에서도 같은 특정 기능을 갖고있는 반면에, spaced multiple CREs* 는 조건에 따라 (context-dependent) 다른 기능을 위한 alternative promoters로 사용된다. 즉, 같은 서열 코드가 polysemy (두개 이상의 역할) 과 distant semantic relationship을 갖고있다고 볼 수 있고 이는 natural langauge 의 주요 특성이다. 이전 언어학 연구들에서 DNA 가 특히 non-coding 부분이 인간의 언어와 많이 유사함을 보인다는 것을 확인하였다 (Brendel and Busse, 1984; Head, 1987; Ji, 1999; Mantegna et al., 1994; Searls, 1992; 2002). 하지만, CREs가 생명학적으로 어떤 의미를 갖고 있는지는 또다른 contexts 로 여전히 많은 부분이 알려지지 않고 있다.
최근, cis-regulatory 부분을 연구하기 위한 유전자 서열 데이터를 이용한 deep learning 부분이 성공적으로 성장하고 있다.
- DNA-protein interactions (Alipanahi et al., 2015)
- chromatin accessibility (Kelley et al., 2016)
- non-coding variants (Zhou and Troyanskaya, 2015)
- CNN-based architecture Zou et al., 2019)
- RNN-based models (LSTM, GRU) (Hochreiter and Schmidhuber, 1997) (Cho et al., 2014)
- hybrids (Hassanzadeh and Wang, 2016; Quang and Xie, 2016; Shen et al., 2018)
* semantic: biological meaning
* cis-regulatory elements (CREs): non-coding DNA that regulates the transcription of neighboring genes. 단백질로 발현이 되지 않는 비암호화 DNA (non-codoing DNA) 부분으로, 이웃 유전체들의 전사/발현을 억제하는 요소이다.
* spaced multiple CREs: 영향을 미치는 gene에서 좀 떨어져있고 두개 이상의 CREs로 작동해야 발현을 작동시킨다.
2. Materials and Methods
DNABERT model 은 NLP 모델인 BERT 와 같은 방식으로 구성된다. BERT 는 tranformer-based* 언어 기반 representation model 이다. DRNBERT 는 처음으로 k 길이의 sequence (k-mer token)을 하나의 sequence set 으로 표현하고 다음과 같이 input 으로 넣는다. 각각의 seqeucne(token) -> 숫자 vector -> embedding -> M matrix 로 표햔된다. 이때 수학적으로는, multi-head self-attention mechanism 을 사용한다.

Tokenization
Tokenization 을 할때, 즉 DNA sequence 를 k-mer로 쪼갤때, k 값에 따라 결과값도 다를 수 있다. 위에서는 k 를 3 으로 설정하였지만 다르게도 설정 할 수 있다. 3-mers: {ATG, TGG, GGC, GCT}, 5-mers: {ATGGC, TGGCT}
Pre-training
DNABERT (max length: 512 bp, mask 설정, training data 생성 (direct non-overlap splitting, random sampling), 120k steps with a batch size of 2000, 12 Transformer layers with 768 hidden units and 12 attention heads in each layer)
Fine-tuning
different k
( + Transformer 을 기반으로 한 대표적인 모델은 seq2seq이 있다. 예시로, 문장 번역을 할때 하나의 sequence (문장) 을 모델을 이용해서 다른 sequence (번역문장)로 transformer 하는 것이다. 보통 encoder(인코더) & decoder(디코더)로 구성되어 있다.
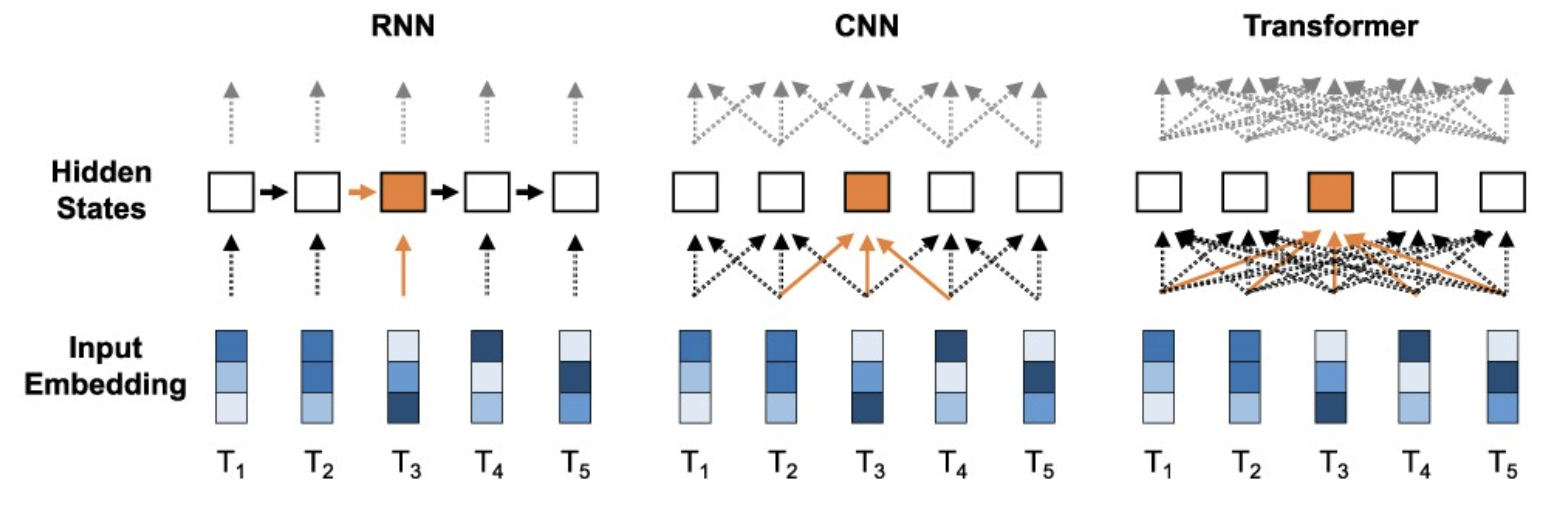
CNN: sequence 의 지역적 특징을 잡아냄
RNN: 시퀀스 정보를 잘 압축. 단어를 차례대로 처리. )
3. Results
1. DNABERT-Prom -> proximal promoter 과 core promoter 부분을 예측할 수 있다.
* proximal promoter: primary regulatory elements 를 포함하는 start position upstream sequence (보통 up to 250 bp)
* core promoter: 전사(transciption)하기위한 최소한의 sequence (e.g. transcription start site; TATA box)
해당 논문에서 두가지 모델로 조절
- DNABERT-Prom-3oo (human TATA box이용)
- DNABERT-Prom-scan (human non-TATA box이용)
2. DNABERT-TF -> transcription factor binding sites 확인
3. DNABERT-viz -> 중요한 regions, context, sequence motifs 시각화
4. DNABERT-Splice -> canonical/non-canonical splice sites
5. functional genetic variants 확인
6. 다른 개체에서의 성능 확인
Yanrong Ji, Zhihan Zhou, Han Liu, Ramana V Davuluri, DNABERT: pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome, Bioinformatics, Volume 37, Issue 15, 1 August 2021, Pages 2112–2120, https://doi.org/10.1093/bioinformatics/btab083


