| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | ||
| 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 13 | 14 | 15 | 16 | 17 | 18 | 19 |
| 20 | 21 | 22 | 23 | 24 | 25 | 26 |
| 27 | 28 | 29 | 30 |
- drug development
- single cell analysis
- javascript
- single cell rnaseq
- cellranger
- 비타민 C
- EdgeR
- github
- Bioinformatics
- scRNAseq analysis
- DataFrame
- python matplotlib
- matplotlib
- scRNAseq
- drug muggers
- single cell
- julia
- ngs
- CUTandRUN
- CSS
- CUT&RUN
- Git
- Batch effect
- HTML
- ChIPseq
- js
- PYTHON
- 싱글셀 분석
- MACS2
- pandas
- Today
- Total
바이오 대표
[ 싱글셀 논문 ] Gene Network 모델 (SCENIC vs WGCNA) “Single-cell network biology for resolving cellular heterogeneity in human diseases” (2020) 본문
[ 싱글셀 논문 ] Gene Network 모델 (SCENIC vs WGCNA) “Single-cell network biology for resolving cellular heterogeneity in human diseases” (2020)
바이오 대표 2023. 7. 22. 03:51Single-cell network biology

Network 찾는 방법
- Bulk RNAseq 에서는 Boolean networks, Bayesian networks, ordinary differential equations (ODEs), information theory, regression, and correlation18,19,20 와 같은 방법이 사용되었습니다.
- scRNAseq 에서도 위의 방법 사용 가능하지만, 싱글셀로만 얻을 수 있는 정보를 이용하기도 합니다. 예로, trajectory analysis (psueotime 정보) 를 기반으로 하는 방법이 있습니다. 즉, 세포들이 특정 developmental/differentiation 상태에 있다고 가정하고 시간대로 나열한, time-ordered transcriptomes 정보를 갖고 regulatory network 추측이 가능합니다. 21,22,23,24 하지만 이는 trajectory analysis algorithm에 아주 의존적이라고 볼수 있습니다. → 근데 pseudotime 정보 없이 진행하는게 좋다고 하는 논문도 있습니다. 25
Network 찾는 방법 모델s (single cell 적용)
** Network Biology - 네트워크의 hub genes을 찾는것에서 부터 시작이라고 볼 수 있습니다.
Boolean models
네트워크 (a set of genes)를 binary states 즉 activated 상태인지 repressed상태인지를 보여주는 모델입니다. 해당접근 방법을 싱글셀에 적용하면, 각 세포를 해당 세포의 유전자패턴을 분석해서, 특정 states에 분류하고 비슷한 세포들끼리 연결하는 방식입니다. 이를 토대로, 중요한 regulators들을 찾을 수 있습니다.
사용 사례: mRNAs 와 protein 간의 상호작용관계 추측에 자주 사용되고, hepatopoiesis (조혈작용)의 GRNs 모델을 만드는데 성공하였습니다.
단점: computational burden이 커서 확장성이 부족합니다. 모델로 만들고 싶은 유전자 100개정도까지만 가능합니다.
Ordinary differential equation (ODE) models
해당 모델은 네트워크의 dynamic에 초점을 둡니다. 따라서 특정 states가 아닌, continous 값을 이용하여 dyanimic system of gene regulation을 예측합니다. 시간에 따른 유전자 발현양 변화를 다른 유전자 (변수)의 영향으로 inhibitory/activating 된다는 점을 고려하여 계산합니다. 현재, nonlinear 을 이용한 방법 중 가장 좋다고 알려져 있습니다.
사용 사례: differentiation과 같이 변하는 상태관련 정보를 얻고 싶을때 사용하기 좋습니다.
Tools: SCODE38
Regression models
대부분의 regeression 을 기반으로 한 네트워크 추측 툴들은 “모든 유전자의 발현양은 간단한 weighted liner equation 의 합으로 표현할 수 있다”라는 가정하에 계산됩니다. 이는 데이터가 independent, residuals(errros)가 normal distribution을 따른다는 조건을 충족해야하는데 싱글셀에는 보통 조건을 충족하지 못합니다. 그래서 싱글셀네트워크를 regression 모델로 표현하기 위해서는 통계적으로 약간 trick(e.g., polynomial modeling, data transformation)을 해서 조건을 충족하도록 해줘야 합니다.
이러한 방식을 사용할때 사용자로써 보통 a list of regulators 즉 transcription factors을 Input 값으로 주면, 알고리즘이 a set of regulators과 특정 타켓 유전자의 발현양의 관계를 해 할 할 수도있 와 주와
Tools: GENIE341 (random forest) , GRNBoost42,43 (좀 더 확장성있고, 빠릅니다)
→ 해당 방식은 bulkRNAseq에 비해 싱글셀에서 진행하면 false-positive links 가 많이때문에 TF binding motif enrichemnt 정보를 이용해서 putative direct-binding targets을 얻을 수도 있습니다. (SCENIC)
Coexpression-based models
보통 Pearson correlation coefficient 나 rank-based Spearman correlation coefficient 계싼을 이용해서 유전자들의 연관성을 파악합니다. 이때는 유전자 발현양의 정확성이 중요하기에 batch correction에도 유의해야합니다.
Gene network databases
STRING50, HumanNet51, and PCNET52
SCINET56 - reference network를 cell-type specific에 맞춰 필터링 가능하게해줍니다.
내가 해당 논문에서 알고 싶었던 것:
SCENIC vs WGCNA.
→ SCENIC은 Transcription factor을 기반으로 네트워크를 형성하고 WGCNA는 유전자의 expression profile을 기반으로 네트워크를 구성합니다.
SCENIC은 TF-regulon subnetworks을 추측하는데 이때, 각 세포는 각 regulon의 activity로 표현하여 계산합니다. 또한, 이를 통하여 세포의 key regulators를 확인하거나, regulon genes의 GSEA을 분석해 생물학적 pathway 해석도 가능합니다. WGCNA (weighted correlation network analysis) 는 거대한 유전자 발현 프로필에서 co-expression network를 추측하는데 많이 사용되는 툴입니다. 이때 유전자에 포커싱보다는 기능적 pathway에 더 포커싱하여 큰 그림을 봅니다. 해당 툴을 다룰때는 특정 parameters들을 사용자가 넣어줘야하는데, 사전 지식이 없으면 힘들 수 있다고 합니다.
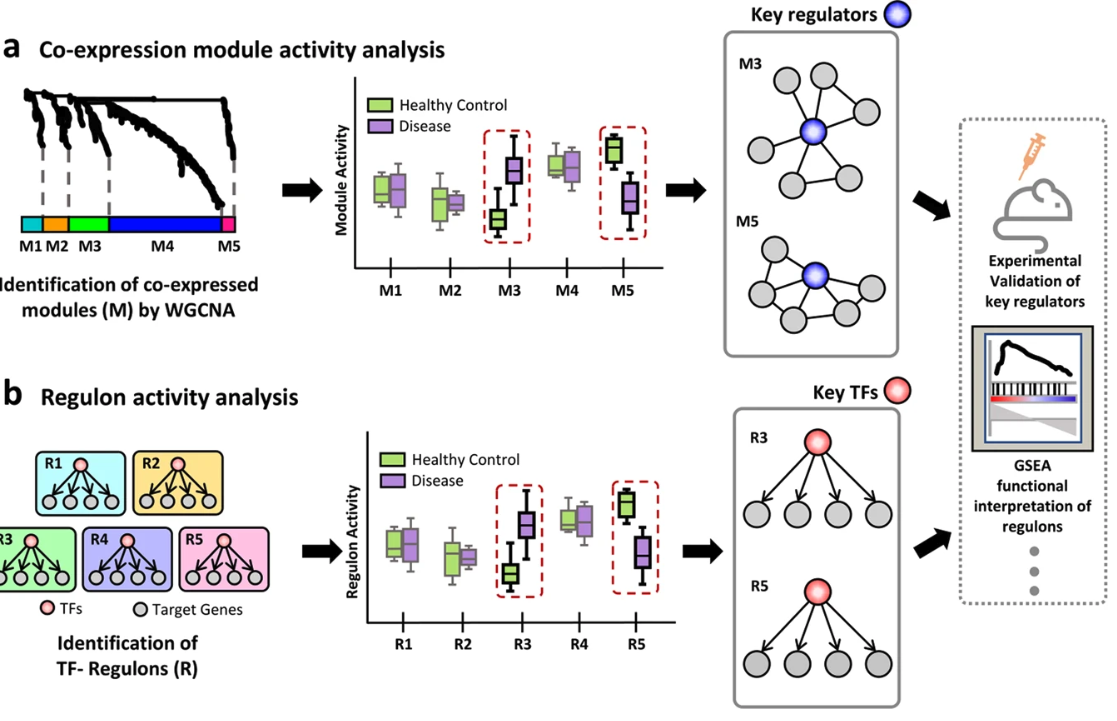
* scWGCNA https://github.com/CFeregrino/scWGCNA
* SCENIC + https://github.com/aertslab/scenicplus
참고
Skinnider, M. A., Squair, J. W. & Foster, L. J. Evaluating measures of association for single-cell transcriptomics. Nat. Methods 16, 381–386 (2019). 에서 gene networks association (correlation, proportionality, similarity) 계산하는 방법 17개 비교. , ϕs,phit and ρp,perb (proportionality) 2개가 제일 좋은 결과를 보였습니다.
** proportionality는 correlation과 비슷하지만 다르게 2개의 feature vector의 dependence 를 계산합니다.




