| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | |||
| 5 | 6 | 7 | 8 | 9 | 10 | 11 |
| 12 | 13 | 14 | 15 | 16 | 17 | 18 |
| 19 | 20 | 21 | 22 | 23 | 24 | 25 |
| 26 | 27 | 28 | 29 | 30 | 31 |
- scRNAseq
- single cell analysis
- Git
- scRNAseq analysis
- ngs
- matplotlib
- EdgeR
- HTML
- 비타민 C
- python matplotlib
- single cell rnaseq
- 싱글셀 분석
- julia
- cellranger
- CSS
- github
- Batch effect
- single cell
- DataFrame
- CUT&RUN
- drug development
- js
- PYTHON
- CUTandRUN
- javascript
- pandas
- MACS2
- Bioinformatics
- drug muggers
- ChIPseq
- Today
- Total
목록분류 전체보기 (216)
바이오 대표
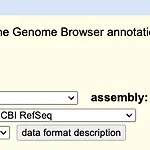 [ IGV ] IGV 에 custom annotation track 만들고 업로드 하기 (유전자 이름으로 검색)
[ IGV ] IGV 에 custom annotation track 만들고 업로드 하기 (유전자 이름으로 검색)
Custom annotation track IGV에서 annotation track을 통해 원하는 유전자 검색으로 해당 위치를 관찰 할 수 있다. 하지만 내가 가지고 있는 데이터가 custom 이여서 유전자 검색이 되지않아서 custom annotation track을 만들어 보았다. How? 보통 “standard’ gene track 은 ncbi refseq 형식이다. 예로, ncbiRefSeq.sorted.txt and ncbiRefSeq.sql (참고 링크와, 그림1,2 참고). Custom genome을 가지고 있다면 annotation track 을 그림2 와 같은 format으로 만들어 줄수 있다. 이는 [1] gtf파일이 있으면 ucsc의 ‘gtfToGenePred’ 로 만들고 [2] co..
 [싱글셀 논문] Background/Ambient RNA 제거 툴 비교 (SoupX, DecontX, Cellbender) “The effect of background noise and its removal on the analysis of single-cell expression data” 2023
[싱글셀 논문] Background/Ambient RNA 제거 툴 비교 (SoupX, DecontX, Cellbender) “The effect of background noise and its removal on the analysis of single-cell expression data” 2023
요약 2개의 다른 subspecies mice (M.m.castaneus 와 M.m.domesticus) 를 이용하여서 background noise 실험을 진행하였다. 사용된 Mice들은 imbred(근친계) 임으로 99% 유전자가 동일하고 homozygous SNP을 이용해서 ~7000 유전자의 subspecies를 구별해 낼 수 있다. 즉, SNP을 이용하여 cell 의 endogenous, exogenous UMI 가 구별이 되고 이를 이용해서 background noise를 계산할 수 있다는 뜻이다. 논문 실험에서는 총 5개의 replicate (3 scRNAseq, 2 snRNAseq)에서 background noise를 계산하였고, 꽤 variability 가 큰 noise 값을 보였다 (3..
DiffBind 를 사용하여 분석하다가, FRiP 스코어가 너무 낮아서 다른 방법을 찾아보았다. Diffbind 에서 계산해주는 FRiP는 consesnsus peak set에 dependent하기 때문에 더 낮은 score을 얻을 수 있다고 한다. FRiP FRiP score 이란 Fraction of Reads in Peaks이다. 말 그대로, 특정 called peaks (region) 에 들어가는 reads의 비율이라고 해석 할 수있다. 이는 usable reads in significantly enriched peaks/total number of mapped reads와 같이 계산된다. 실제 sample의 FRiP 계산해보기 1. 맵핑된 sequencing data (BAM/SAM) 과 cal..
Python package “Click: command line interface creation kit“ Click 패키지는 command line interfaces를 이쁘게 작성할 수 있게 도와주는 툴이다. 또한 CLI API 에서 생기는 문제들을 예방하고 보다 빠르고 재미있게 코드를 짤수있도록 도와주고 크게 3가지 장점이 있다. arbitrary nesting of commands help page 를 자동생성 supports lazy loading of subcommands at runtime # test.py 코드 import click @click.command() @click.option('--count', default=1, help='Number of greetings.') # help..
