| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | ||||
| 4 | 5 | 6 | 7 | 8 | 9 | 10 |
| 11 | 12 | 13 | 14 | 15 | 16 | 17 |
| 18 | 19 | 20 | 21 | 22 | 23 | 24 |
| 25 | 26 | 27 | 28 | 29 | 30 | 31 |
- js
- Bioinformatics
- PYTHON
- javascript
- HTML
- CSS
- pandas
- single cell analysis
- Git
- EdgeR
- CUT&RUN
- 싱글셀 분석
- python matplotlib
- DataFrame
- drug muggers
- MACS2
- ChIPseq
- drug development
- single cell
- single cell rnaseq
- matplotlib
- cellranger
- 비타민 C
- CUTandRUN
- Batch effect
- ngs
- julia
- scRNAseq
- scRNAseq analysis
- github
- Today
- Total
목록Bioinformatics (19)
바이오 대표
 [SEACR 논문] “Peak calling by Sparse Enrichment Analysis for CUT&RUN chromatin profiling”
[SEACR 논문] “Peak calling by Sparse Enrichment Analysis for CUT&RUN chromatin profiling”
Background DNA 의 어느 부분에 protein 이 붙어있는지에 따라, 유전자 발현양이나, 세포의 운명? 이 결정된다. 따라서 해당 위치를 알아내기 위해 사용되는 기술로는 ChiP-seq(protein 과 dna 를 link 하고 dna 자른 뒤 antibody로 추출하는) 과 CUT&RUN(Antibody-directed MNase 가 protein binding 위치를 자르는 기술) 이 있다. ChiP-seq 기술이 좀더 오래 사용되었고, 많은 분석 툴들이 해당 기술에 초점이 맞춰져있어서 이를 CUT&RUN 데이터에 적용시키기 어려운면이 있다. Chip-seq 데이터는 background noise 가 큰 반면, CUT&RUN low read length, low and sparseness ..
 [ 싱글셀 분석 ] 10x Cell ranger 정복하기 1
[ 싱글셀 분석 ] 10x Cell ranger 정복하기 1
Cell Ranger 이란? (v7.1) Illumina 의 Chromium single cell data 를 align 하고 feature-barcode matrics를 만들고, clustering secondary analysis 등을 하기 위한 분석 파이프라인 set 입니다. Cell ranger을 이용해서 데이터를 핸들링 할때 크게 다음 5개의 파이프라인 사용이 가능합니다. cellranger mkfastq Demultiplexing BCL(raw base call) files → FASTQ files bcl2fastq cellranger count Illumina single cell FASTQ가 주어진다면 가장 흔하게 사용되는 파이프라인입니다. Alignment, filtering, barco..
 [ Cut & Tag / Cut & Run ] Cut & Tag 투토리얼
[ Cut & Tag / Cut & Run ] Cut & Tag 투토리얼
해당 글을 https://yezhengstat.github.io/CUTTag_tutorial/#Cite_this_tutorial 을 참고하였습니다. CUT & TAG Data Processing and Analysis Tutorial 1. Introduction Eukaryotic nucleus DNA에서 일어나는 모든 dynamic process 는 chromatin landscape/지형 을 따른다고 보면된다. Chromatin landscape 는 nucleosomes, their modification, TF and chromatin-associated complexed 등으로 이루어져있다. 이렇게 다는 chromatin features 을 이용하면 다른 cell type간, development..
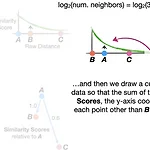 [single cell Analysis] 싱글셀 분석 기본 다지기 4 - 클러스터 visualization (UMAP)
[single cell Analysis] 싱글셀 분석 기본 다지기 4 - 클러스터 visualization (UMAP)
- PCA - 해당 글에서는 UMAP 관련해서 좀 자세히 다뤄보려 한다. 다음 내용은 StatQuest 을 참조하였다. UMAP 을 이용해서 High dimension 데이터를 low-dimension에 표현할 수 있다. How? 전체적인 그림은, 낮은차원에서 point를 움직여서, high dimention 에서와 비슷한 모습을 보이도록 조정하는 방법이고 이를 Similarity score 을 계산하여 사용한다. 1. High-dimention points 에서 서로간의 distance 를 계산한다. 2. High-dimention neighbor 숫자 (default 15) 에 따라 log2 (#of neighbor) 을 이용하여 curve 를 그리고, 각 포인트의 similarity score 을 ..
